简介
KSEA(Kinase Substrate Enrichment Analysis)是一个用于分析激酶与其底物之间相互作用的工具,可以帮助研究者探索激酶活性的变化。KSEA提供了在线网站和R语言程序包两种使用方式。
主要特点
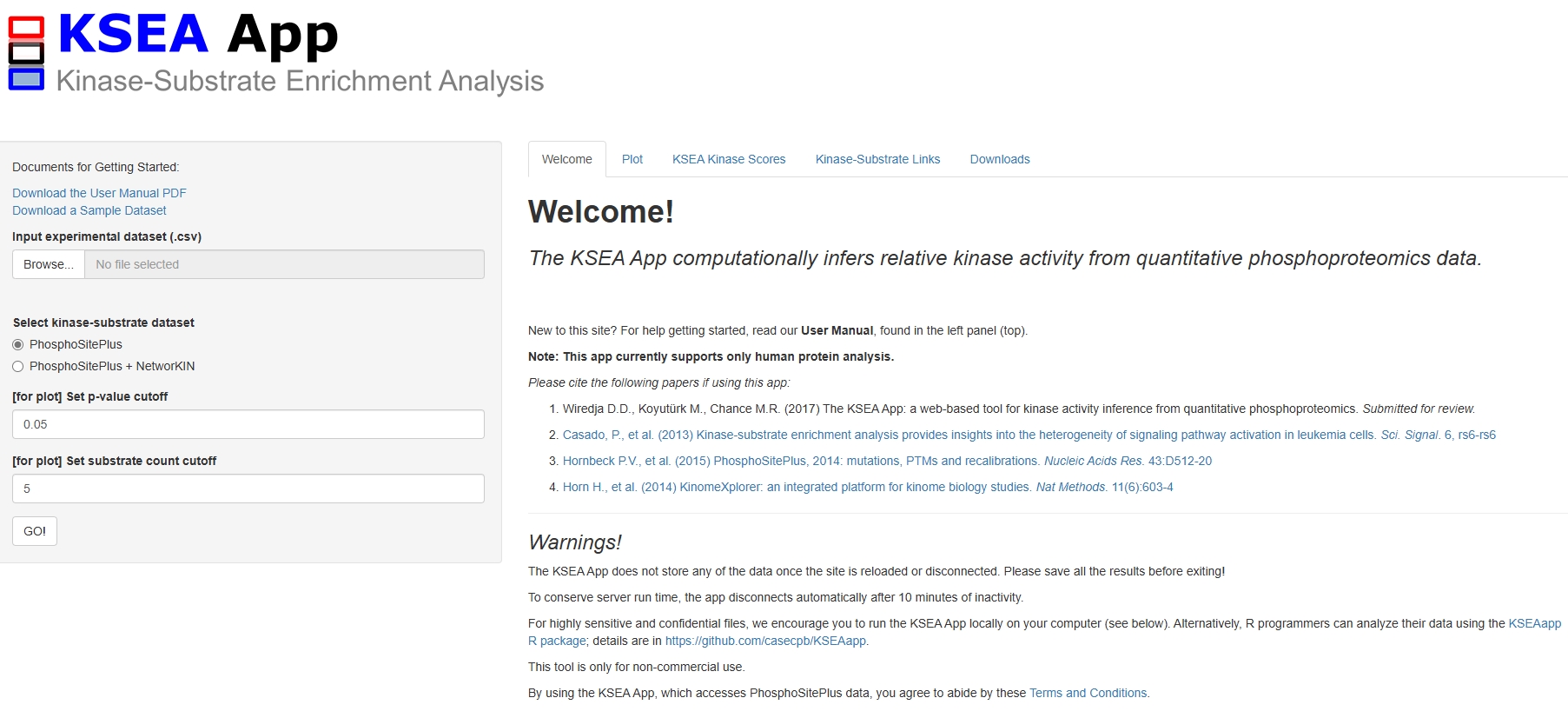
在线运行和R语言程序包:用户可以通过在线网站直接运行KSEA,也可以下载R语言程序包进行分析。
上传数据集:用户需要上传待分析的激酶底物数据集进行分析。
选择数据库:在线网站提供了激酶底物数据库注释,用户无需上传注释文件。
设置分析阈值:用户可以设置分析的阈值,以调整结果的严格性。
快速获得结果:点击“GO”后,KSEA会快速完成分析并展示结果。
主要结果
Kinase-Substrate Links:展示激酶和底物的对应关系、数据库来源和倍数变化等信息。
KSEA Kinase Scores:记录激酶的富集得分、z-score和显著性p值,用于推断激酶活性。
可视化:通过“Plot”功能,可视化激酶的富集结果,根据z-score值排序,并用颜色标注显著的活性变化。
下载结果:用户可以下载富集分析的结果表格和图片,方便本地查看和后续分析。
使用场景
生物医学研究:用于研究激酶活性在疾病发生、发展中的作用。
系统生物学:探索细胞信号传导网络中激酶的调控作用。
药物发现:分析药物对激酶活性的影响,辅助药物靶点的发现。
结论
KSEA作为一个专门用于分析激酶底物相互作用的工具,为用户提供了一个简便、快速的分析途径。无论是通过在线网站还是R语言程序包,用户都能够轻松地进行激酶活性分析。KSEA的结果不仅提供了激酶和底物的详细信息,还包括了富集得分和显著性分析,以及直观的可视化展示,极大地方便了科研人员对激酶活性变化的研究。通过下载功能,用户还可以将结果用于进一步的本地分析和报告撰写。